少し間が空きましたが、前回の投稿(Rによる主成分分析)ではggbiplotを用いて主成分分析結果を作図しました。今回はやり方を変えて、グループごとに枠で囲む方法を説明します。使用するデータは前回と同様です。
データの準備
プロットする前にデータを少し編集する必要があります。前回に引き続き、xという変数にアヤメのデータ、pcaという変数にその主成分分析結果が入っているとします。以下にそれぞれの要約を示してみます(図1)。
 |
| 図1 |
次にプロット用データを作成し、変数df.pcaへ代入します。今回は第一主成分と第二主成分でプロットするとします。pcaから第一主成分と第二主成分の主成分得点、xからそれぞれの行に対応する種名を抜き出してデータフレームにまとめています。ここで、左辺のPC1、PC2、Speciesは任意です。[]内の数字を3や4にすることで、第三主成分、第四主成分を抜き出すこともできます。
df.pca <- data.frame(PC1=pca$x[,1], PC2=pca$x[,2], Species=x$Species)
グループ分け
グループ分けにはdplyrライブラリを使います。なければインストールしてください。
library(dplyr)
hull <- df.pca %>%
+ dplyr::group_by(Species) %>%
+ dplyr::do(.[chull(.[c('PC1','PC2')]),])
少々ややこしいですが、打ち間違えないよう注意してください。
プロット
プロットにはggplot2を用います。まずデフォルトでプロットしてみましょう。一度プロットを変数pへ代入してから表示します。
library(ggplot2)
p <- ggplot(df.pca, aes(x = PC1, y = PC2, shape = Species)) + geom_point()
pこれで以下のような図が表示されると思います(図2)。
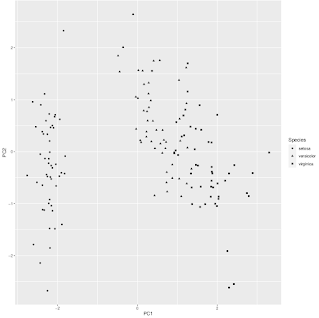 |
| 図2 |
ggplot2の良いところはこの変数pに書き加えていくことで図を編集することができることです。もっと表示をシンプルにしてみましょう(図3)。
p + theme_test()
 |
| 図3 |
次にプロットのシンボルを変更します(図4)。
p + theme_test() + scale_shape_manual(values=c(1,2,3))
 |
| 図4 |
このような感じで図を編集することが可能です。一度ここまでの編集内容を変数p2へ保存しておきます。
p2 <- p + theme_test() + scale_shape_manual(values=c(1,2,3))
その他、編集方法についての詳細はggplot2のドキュメントを参照してください。
各グループを枠線で囲う
それではこれまで作った図に枠線を追加します。グループ分けのところで作成した変数hullを利用します(図5)。なお、alphaは枠内の塗りつぶしの透過度で0が透明です。colorは枠線の色を示します。1は黒です。
p2 + geom_polygon(data=hull, alpha=0, color=1)
 |
| 図5 |
最後に、全ての枠線が実線というのもアレなので、グループごとに線の種類を変えましょう(図6)。
p2 + geom_polygon(data=hull, alpha=0, color=1, aes(linetype=Species))
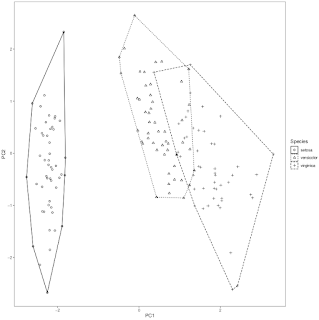 |
| 図6 |
以上です。




0 件のコメント:
コメントを投稿